Diseño y estandarización de PCR cuantitativa en Tiempo Real del virus Madariaga


Autores/as
DOI:
https://doi.org/10.37980/im.journal.rmdp.20252479Palabras clave:
arbovirus, alfavirus, zoonótico, virus Madariaga, RT-PCRResumen
Introducción: Madariaga (MADV) es un virus de ARN de la familia Togaviridae, género Alfavirus. MADV es un virus zoonótico endémico en Panamá, y se asocia principalmente con encefalitis en equinos, y una alta letalidad que va desde el 30-50%. Desde su aparición en humanos en el 2010, ha ocasionado brotes de enfermedad febril y neurológica, especialmente en niños. El diagnóstico de este virus, tanto en humanos como en animales, es limitado por la escasez de metodologías disponibles y actualmente no hay un método para detección de la carga viral. Material y método: Utilizando secuencias publicadas, se diseñó una técnica de RT-qPCR en tiempo real específica para la cuantificación de MADV, que fue validada y estandarizada con aislados de virus panameños de diferentes años. Resultados: La técnica desarrollada es específica y no detecta otros alfavirus presentes en Panamá, y tiene un límite de detección de 100 copias de ARN viral por mL. Conclusión: Esta metodología permite evaluar la carga viral en muestras humanas, de animales y de vectores; siendo una herramienta que podrá ser utilizada para futuros estudios en virulencia, patogénesis y respuesta inmune, y el ciclo de transmisión de este virus en vectores.
Introduction: Madariaga (MADV) is an RNA virus of the Togaviridae family, genus Alfavirus. MADV is a zoonotic virus endemic in Panama, and is mainly associated with encephalitis in equines, and a high lethality ranging from 30-50%. Since its appearance in humans in 2010, it has caused outbreaks of febrile and neurological disease, especially in children. The diagnosis of this virus, both in humans and animals, is limited by the scarcity of available methodologies and there is currently no method for detection of viral load. Materials and method: Using published sequences, a specific real-time RT-qPCR technique for MADV quantification was designed, validated and standardized with Panamanian virus isolates from different years. Results: The developed technique is specific and does not detect other alphaviruses present in Panama and has a detection limit of 100 copies of viral RNA per mL. Conclusion: This methodology allows evaluating the viral load in human, animal and vector samples; being a tool that could be used for future studies on virulence, pathogenesis and immune response, and the transmission cycle of this virus in vectors
El virus Madariaga (MADV) conocido anteriormente como el virus de la Encefalitis Equina del Este Suramericano (EEEV-SA) pertenece a la familia Togaviridae, género Alfavirus, con un genoma ARN de un tamaño aproximado de 10 a 12 kb [1]. MADV es un arbovirus endémico en Panamá asociado con brotes de encefalitis en caballos y otros equinos desde aproximadamente 1936 cuando fue documentado por primera vez en el país [2]. Estudios epidemiológicos en Panamá y América Latina, han intentado establecer asociación entre la infección de MADV y la enfermedad en humanos durante varias décadas, con resultados sugiriendo la avirulencia de este virus y su limitada capacidad para causar enfermedad humana. Sin embargo, recientemente en el año 2010, se ha reportado la emergencia de MADV como un patógeno humano en Panamá [3].
El ciclo de transmisión aún no es completamente claro, sin embargo, se sospecha que los mosquitos del género Culex actúan como su principal vector [4], específicamente las especies C. melanoconion, C. taeniopus, C. pedroi y C. bidens [2,5]. Además, hay evidencia de que los roedores podrían desempeñar un papel como reservorios de este virus, siendo las especies Zygodontomys brevicauda, Oryzomys coesi y Transandinomys bolivaris las más relevantes [6,7]. Uno de los principales retos en los países tropicales es la circulación simultánea de diversos arbovirus, muchos de los cuales comparten síntomas similares, lo que dificulta su diagnóstico clínico [8]. La detección de MADV en humanos se ha dado principalmente durante brotes de encefalitis en equinos y en estudios de vigilancia durante epidemias de otros arbovirus, como se evidenció en Haití y Brasil [9,10,11]. Además, investigaciones de seroprevalencia en Perú [12] y Panamá [13] han identificado anticuerpos contra MADV en personas asintomáticas sugiriendo que podría estar pasando desapercibido o confundido con otras enfermedades en los sistemas de salud [14].
Los síntomas más comunes observados en los recientes casos documentados de humanos con MADV son fiebre, vómitos y convulsiones [15]. No todos los casos desarrollan encefalitis; algunos pueden manifestarse como un síndrome febril indiferenciado [9,11]. Sin embargo, cuando se presenta la encefalitis, generalmente culmina en enfermedad neurológica grave con secuelas permanentes [3]. El diagnóstico de MADV durante el desarrollo de este estudio, en muestras agudas (0-5 días) se basaba en reacción de cadena de polimerasa (PCR) en tiempo final genérica y anidada, seguida de secuenciación por método de Sanger [10, 11]. Recientemente se ha implementado una técnica de tiempo real que acorta el tiempo de respuesta [16]. Para muestras convalecientes (6 días en adelante) se sigue utilizando el ensayo inmunoabsorbente (ELISA) casero, y para diferenciar anticuerpos específicos neutralizantes, se aplica la técnica de Neutralización en placa (PRNT). Estas técnicas requieren de personal altamente capacitado, tanto en biología molecular como en virología tradicional, así como infraestructura especializada y recursos financieros. Estos factores, en conjunto, dificultan su diagnóstico.
La razón de la aparición de MADV y su afectación en humanos desde el 2010 no es clara. Sin embargo, desde entonces se han observado casos esporádicos en Panamá y en otros países de América Central, Sudamérica y, más recientemente, en el Caribe [3, 17, 9]. Luego de una revisión bibliográfica en busca de metodologías moleculares no encontramos RT-qPCR específicas para MADV o que permitan cuantificar la carga viral. Las RT-PCR publicadas en el momento del estudio solo utilizaban la variante del norte en su diseño [18-21]. lo cual podría disminuir la sensibilidad de la detección de MADV ya que existen suficientes diferencias genéticas bien documentadas entre ambos virus que dieron a lugar ser clasificadas como dos especies virales distintas [5].
Por otro lado, existe evidencia científica que relaciona una alta carga viral con un desarrollo más agresivo de la enfermedad, complicaciones graves y hospitalizaciones prolongadas [22-25]. Dado que Panamá es uno de los principales países con reporte de casos y afectados por este virus, este trabajo tiene como objetivo desarrollar una herramienta cuantitativa específica para detectar y medir las partículas virales de MADV. Esta técnica contribuirá a la investigación sobre la transmisión, la virulencia, patogénesis y respuesta inmune del virus, áreas poco estudiadas, y permitirá comprender mejor su tropismo celular, su relación con enfermedades graves y la respuesta inmune del hospedador.
Este es un estudio de tipo experimental, in vitro que fue registrado en la plataforma RESEGIS del Ministerio de Salud de Panamá bajo el número 3451 y fue exento de revisión de protocolo por el Comité de Bioética del Instituto Conmemorativo Gorgas Estudios de la Salud (ICGES) Nº169/CBI/ICGES/19 ya que no se utilizaron muestras o datos humanos. Las muestras de este estudio fueron aislados virales de Madariaga, Chikungunya, Encefalitis Equina Venezolana y Mayaro conservados en el repositorio de cepas virales del Departamento de Investigación de Virología y Biotecnología (DIVB) del ICGES, preservados en congeladores a -80°C. Todos los experimentos en donde se utilizó virus infeccioso fueron realizados en las instalaciones del laboratorio de bioseguridad de riesgo biológico nivel 3 (BSL-3) del ICGES.
Diseño de oligonucleótidos específicos
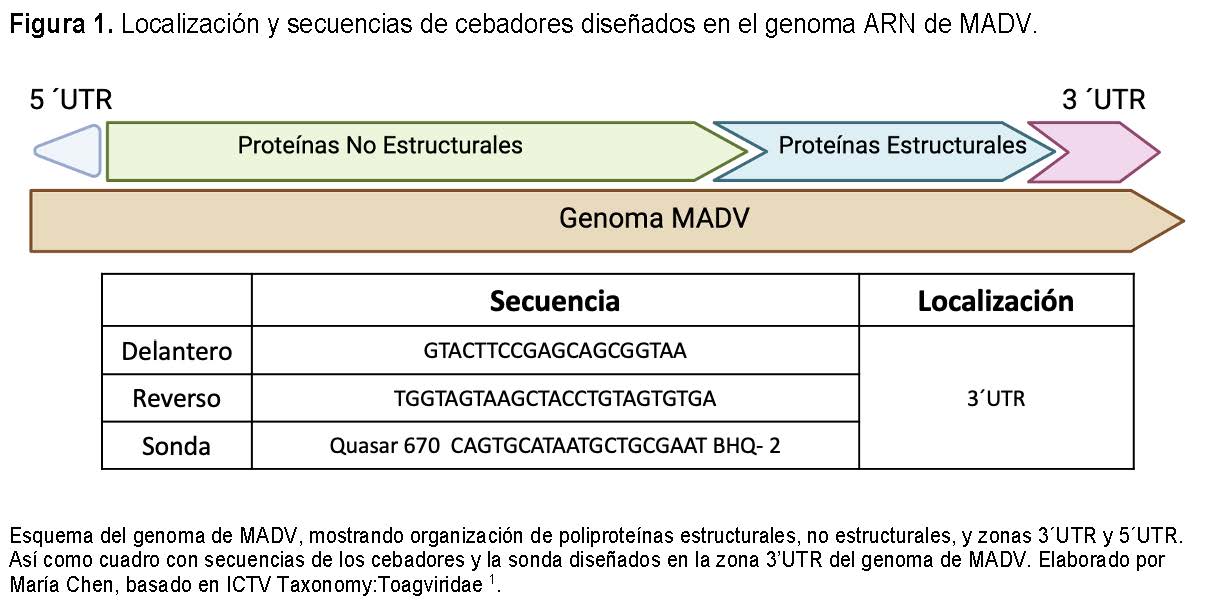
Se descargaron 32 secuencias completas del genoma de MADV desde las plataformas GenBank y la base de recursos de análisis de virus patógenos (ViPR). Estas fueron agrupadas por similitud, dando un subgrupo de 23 secuencias que fueron utilizadas para este trabajo (ver Tabla Suplementaria 1). Estas secuencias fueron exportadas al programa Sequencher® (versión 5.4.6), donde se alinearon para generar una secuencia consenso. A partir de esta secuencia consenso, utilizando el programa en línea PRIMER 3, se diseñaron juegos de cebadores y sondas que reconocen las regiones conservadas del genoma de MADV. Los cebadores y sondas fueron analizados in silico con el software Oligo Evaluator (Sigma Aldrich) para evaluar su composición y detectar posibles problemas como la formación de dímeros o estructuras secundarias, terciarias o cuaternarias. La especificidad de los cebadores y sondas fue verificada mediante alineamientos nucleotídicos utilizando la herramienta BLAST. Finalmente, estos cebadores y sondas fueron sintetizados por la compañía LGC, Biosearch Technologies, en Estados Unidos.
Estandarización de la PCR en tiempo real para MADV
Producción de controles
Los aislados panameños utilizados fueron MADV1962 (GenBank KJ469565), MADV1984 (GenBank GU001936) y MADV2017 (GenBank OR644805), de los cuales se realizó una producción a gran escala.
Para esto se utilizaron células VERO (células de riñón de mono verde africano, ATCC-CCL-81) que fueron infectadas con cada aislado de MADV en el laboratorio de bioseguridad nivel 3 (BSL3) del ICGES. La infección se realizó con una hora de absorción a 37°C y 5% de CO2, utilizando Medio Esencial Mínimo (MEM) suplementado con 2% de Suero Fetal Bovino (SFB), 0.5% de Anfotericina y 1% de Penicilina/Estreptomicina. Las células se monitorearon cada 24 horas bajo un microscopio invertido (Nikon Eclipse TS100, objetivo 10x 0.25 Ph1ADL) hasta que se observó un efecto citopático (CPE). Cuando aproximadamente el 75% de las células mostraron CPE, se procedió a cosechar la producción de cada virus. Posteriormente, se extrajo el ARN viral de cada aislado utilizando el kit QIAamp viral RNA mini kit (Qiagen, catálogo 52906), siguiendo las instrucciones del fabricante.
Determinación de la concentración óptima de cebadores y sonda
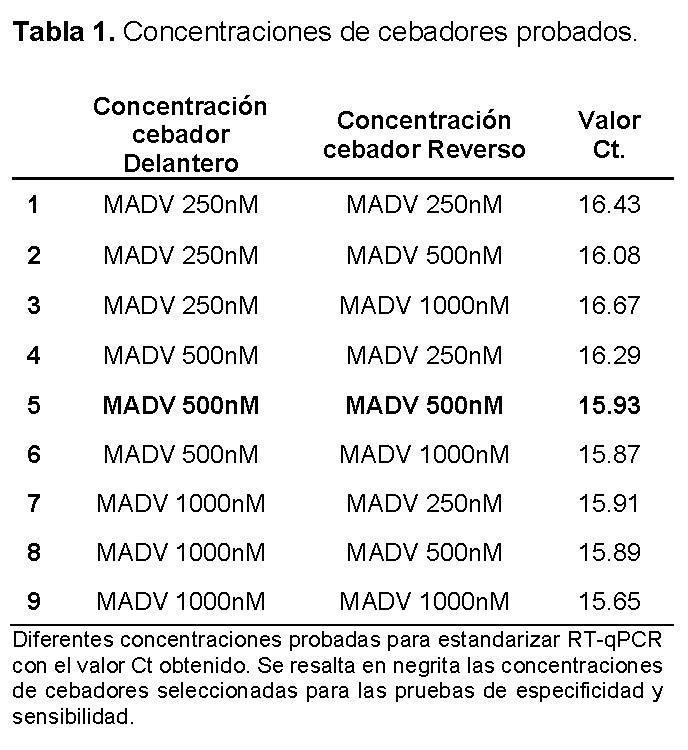
Utilizando el kit SuperScript™ III First-Strand Synthesis System (Invitrogen, catálogo 18080051) se sintentizó ADN complementario de primera cadena a partir del ARN de MADV. Se procedió a probar distintas concentraciones de los cebadores (250, 500 y 1000 nM) con el kit de SYBR Green PCR Master Mix (Thermo Fisher Scientific, catálogo 4309155) utilizando el ADNc. De igual manera, para la sonda se probaron las concentraciones de 100, 150, 200, 300 y 400 mM con el kit SuperScript™ III Platinum™ One-Step qRT-PCR (Invitrogen, catálogo 11732088) directamente a partir del ARN viral. Estas pruebas fueron llevadas a cabo con los ARNs de los aislados controles de MADV, en un volumen final de reacción de 25 uL en el equipo Applied Biosystems 7500Fast (Thermo Fisher Scientific).
Prueba de especificidad
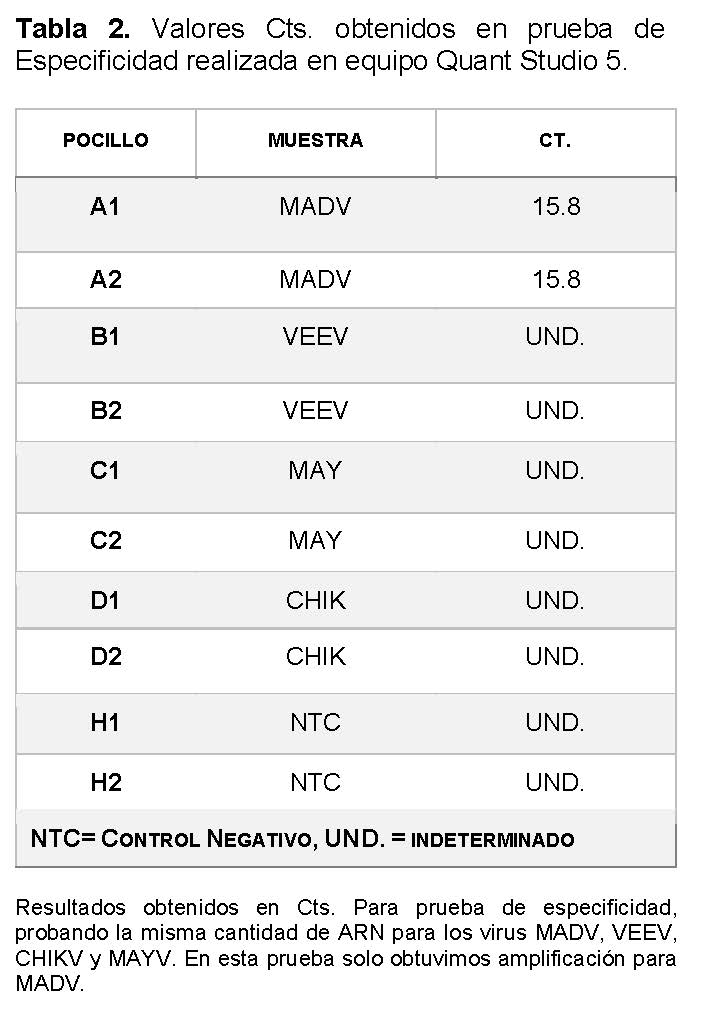
Una vez encontrada la mejor concentración tanto de sondas como cebadores, se realizó un ensayo para comprobar su especificidad, utilizando ARN viral de los virus MADV, Encefalitis Equina Venezolana (VEEV) (ICGES_A001877), Chikungunya (CHIKV) (ICGES_256137) y Mayaro (MAYV) (AVR0565), todos del género Alfavirus, en el equipo Quant Studio 5 (Applied Biosystems).
Construcción de plásmido MADV para cuantificación
Para la construcción del plásmido MADV se utilizó el vector comercial pGEM TEasy-Vector (Promega, catálogo A1360) en el cual se insertó el fragmento amplificado de la PCR obtenido con el aislado MADV1984 (Figura suplementaria 1). El plásmido final fue verificado por medio de digestión enzimática, utilizando la enzima ECORI (NEB R0101s) y por medio de secuenciación de Sanger utilizando los cebadores M13.
Límite de detección de la técnica de RT-qPCR
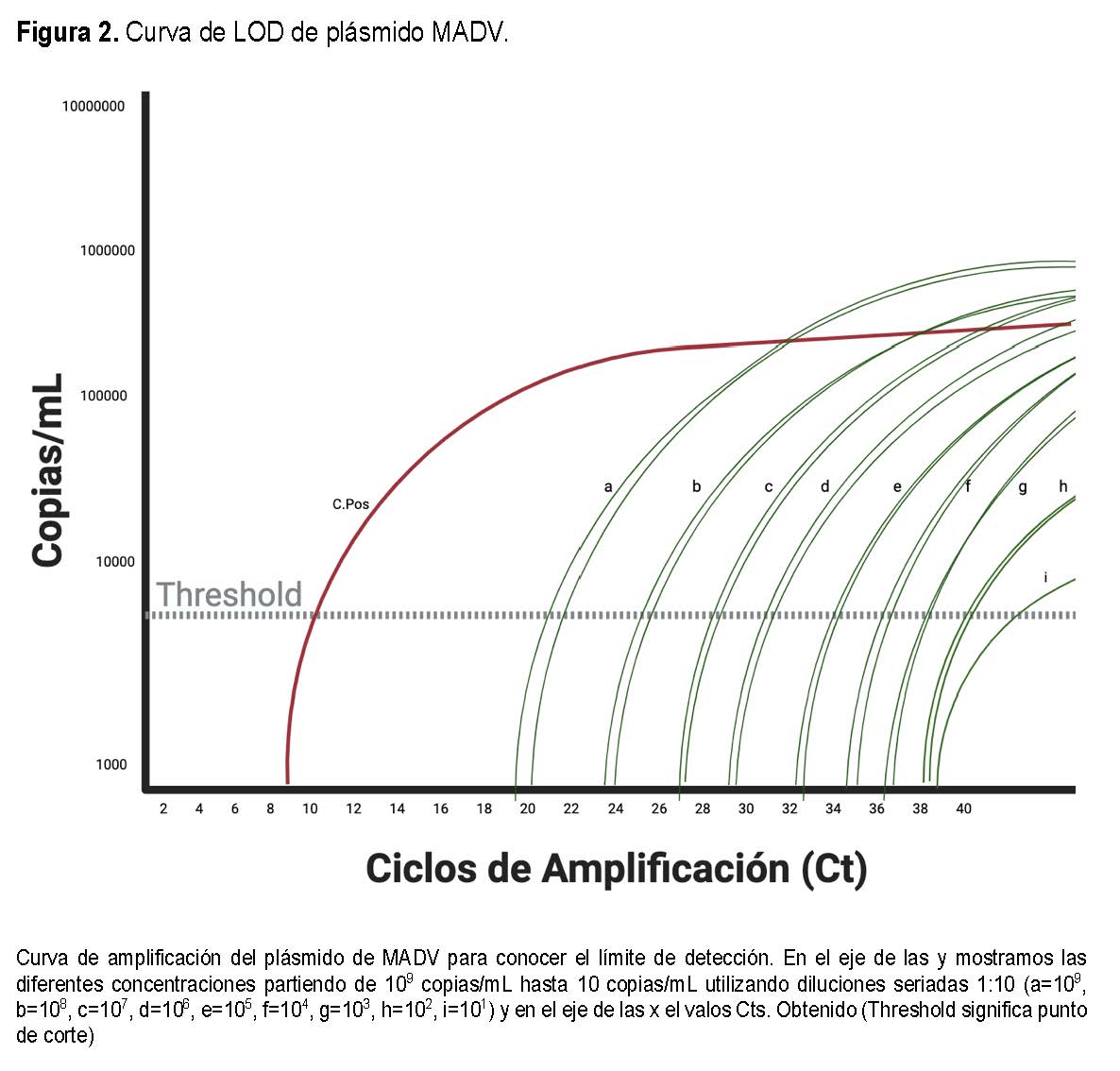
El ADN del plásmido construido se cuantificó en el NanoDrop 200c (Thermo Scientific) y se determinó el número de copias por mL. Para conocer el límite de detección (LOD por sus siglas en inglés límite de detección) de la técnica RT-qPCR diseñada, se procedió a hacer diluciones seriadas del plásmido con un fragmento de MADV desde la concentración 109 hasta 10 copias/mL. Cada dilución se hizo en duplicado y se añadió un pocillo de control negativo y otro positivo en el termociclador de tiempo real QuantStudio5 (Applied Biosystems™) con las condiciones previamente estandarizadas.
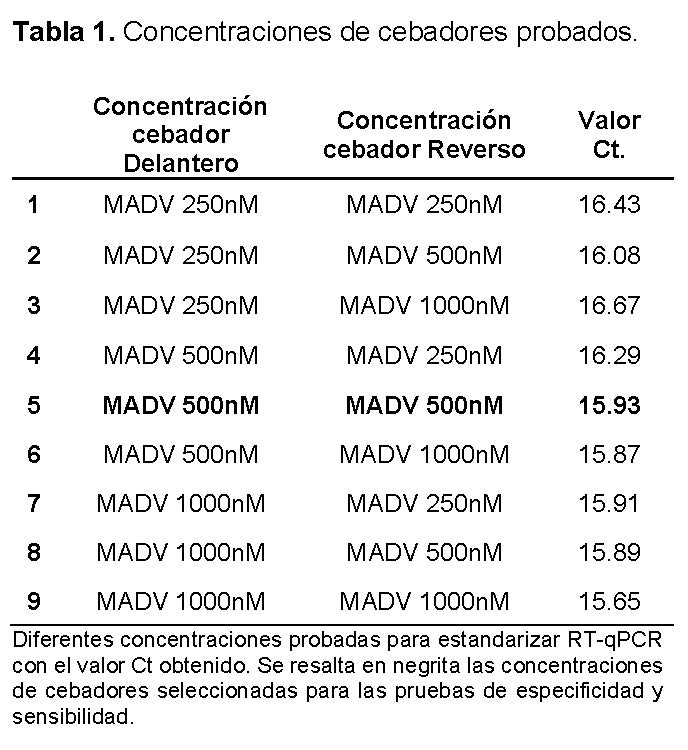
Para detectar especificamente a MADV, se diseñó un set de cebadores y sonda que detectan la región 3´UTR, amplificando un fragmento de 111 nucleótidos (nt) (Ver Figura 1). Para estandarizar la metodología se utilizó como matriz el ARN viral del aislado de MADV del 2010. Los resultados se visualizaron por las curvas de fusión, curva de amplificación y los valores de corte (Cts.). Se consideró la mejor concentración para cebadores y sonda, aquella que permitió obtener una buena señal de fluorescencia asociada a una buena sensibilidad y a su vez permite optimizar el uso de reactivos. Se escogió la concentración de 500 nM para los cebadores, tanto delantero como reverso, y la concentración de 200 nM para la sonda (Tabla 1).
Para el análisis de la especificidad, se obtuvo amplificación para el aislado de MADV, con los parámetros de los cebadores y sonda seleccionados durante la implementación de la técnica, pero no para los virus VEEV, CHIKV o MAYV que son virus del mismo género Alfavirus (Ver Tabla 2). Como no se contaba con el virus de la Encefalitis Equina del Este (EEEV-NA) que es el Alfavirus con genoma con mayor similitud a MADV, y no fue posible importarlo debido a regulaciones de bioseguridad y biocustodia por su clasificación como agente selecto en Estados Unidos que es donde mayormente circula; se realizaron pruebas de amplificación in silico con el programa Genius Prime utilizando una secuencia de referencia de EEEV-NA (NC_003899) con los oligonucleóticos y sondas diseñados para MADV. En esta simulación in silico de especificidad, no hubo amplificación.
Para determinar el límite de detección (LOD) de la prueba, se realizó una curva de dilución del plásmido que contiene el fragmento de MADV, y se logró cuantificar hasta 100 copias/mL(ver Figura 2), con un corte de positividad de Ct de 38 (ver Tabla 3).
En este estudio, se desarrolló y estandarizó una RT-qPCR cuantitativa en tiempo real para la detección de MADV, un virus emergente en Panamá y otras regiones de las Américas. Esta técnica reconoce la región 3'UTR del genoma viral, que contiene elementos cruciales para la replicación viral y sitios de unión para los microARN celulares, los cuales son clave en la determinación del tropismo celular, el rango de huéspedes y la patogénesis del virus [26].
A pesar de que la región escogida es altamente conservada, no tuvimos reacción cruzada con virus del mismo género como VEEV, CHIKV o MAYV ya que poseen diferencias suficientes entre nucleótidos para no amplificar la región seleccionada durante el diseño de la prueba. Una de nuestras limitaciones fue que no pudimos probar esta técnica in vitro con el EEEV-NA, sin embargo, la prueba in silico realizada con el programa Genius Prime no obtuvo amplificación, mostrando una gran especificidad de la prueba hacia MADV.
La RT-qPCR diseñada logró detectar hasta 100 copias de ADN plasmídico conteniendo un fragmento de MADV/mL, lo que representa una LOD 10 veces mayor a la PCR anidada en tiempo final que fue utilizada durante varios años como metodología diagnóstica y posee un LOD de 1000 copias en su PCR inicial [27].
Teniendo en cuenta que el objetivo principal de este estudio era obtener una metodología rápida y cuantitativa para estudios de investigación en virología e inmunología de MADV, es importante señalar que la estandarización fue realizada con ARN viral procedente de aislados producidos en cultivos de células de primates no humanos infectadas, no se probaron ni con mosquitos ni con muestras veterinarias ni con muestras clínicas y por esta razón podría haber variabilidad en la sensibilidad de esta técnicas con diferentes tipo de muestras provenientes de diferentes especies.
A pesar de que, al inicio de este estudio, no existían técnicas moleculares específicas para la detección de MADV, en 2023 se optimizó una RT-qPCR multiplex validada tanto para mosquitos como para muestras humanas, la cual permite diferenciar entre el virus de Encefalitis Equina Venezolana (VEEV) y MADV [16]. Esta nueva técnica ha reducido el tiempo de respuesta en el diagnóstico diferencial de las encefalitis. Sin embargo, la RT-qPCR de MADV desarrollada en este estudio ofrece, además de la identificación del virus, la capacidad de cuantificar cargas virales por medio del plásmido construido.
La técnica de RT-qPCR en tiempo real desarrollada permite la cuantificación específica del MADV con un límite de detección de 100 copias de ARN viral por mL. Este trabajo abre la puerta a futuros estudios en biomedicina, como la caracterización de la respuesta inmune a nivel celular, la búsqueda de antivirales o posibles blancos terapéuticos en los cuales será crucial poder cuantificar la replicación viral.
Agradecimientos
Agradecemos a la Mgter. Yamitzel Zaldivar y al personal del Departamento de Investigación y Vigilancia de Riesgo Biológico 3 del ICGES por entrenamientos y soporte en el uso del laboratorio BSL3, a Briggite Henríquez y Angélica Castro por asistencia técnica con el secuenciador Sanger, a los doctores Davis Beltrán y Celestino Aguilar por asistencia en la elaboración de imágenes y análisis bioinformáticos.
Referencias
[1] Chen R, Mukhopadhyay S, Merits A, et al. ICTV virus taxonomy profile: Togaviridae. J Gen Virol. 2018;99(6):761-762. doi:10.1099/jgv.0.001072
[2] Dietz WH Jr, Galindo P, Johnson KM. Eastern equine encephalomyelitis in Panama: the epidemiology of the 1973 epizootic. Am J Trop Med Hyg. 1980;29(1):133-140. doi:10.4269/ajtmh.1980.29.133
[3] Carrera JP, Forrester N, Wang E, et al. Eastern equine encephalitis in Latin America. N Engl J Med. 2013;369(8):732-744. doi:10.1056/NEJMoa1212628
[4] Stechina OS, Oria GI, Torres C, Diaz LA, Contigiani M, Stein M. First detection of Madariaga virus in mosquitoes collected in a wild environment of northeastern Argentina. Am J Trop Med Hyg. 2019;101(4):916-918. doi:10.4269/ajtmh.19-0475
[5] Arrigo NC, Adams AP, Weaver SC. Evolutionary patterns of eastern equine encephalitis virus in North versus South America suggest ecological differences and taxonomic revision. J Virol. 2010;84(2):1014-1025. doi:10.1128/JVI.01586-09
[6] Vittor AY, Armien B, Gonzalez P, et al. Epidemiology of emergent Madariaga encephalitis in a region with endemic Venezuelan equine encephalitis: Initial host studies and human cross-sectional study in Darien, Panama. PLoS Negl Trop Dis. 2016 ;10(4):e0004554. Doi :10.1371/journal.pntd.0004554
[7] Carrera JP, Galé J, de Souza WM, et al. Madariaga and Venezuelan equine encephalitis virus seroprevalence in rodent enzootic hosts in Eastern and Western Panama. bioRxivorg. Published online August 29, [2023] doi:10.1101/2023.08.28.555226
[8] Aguilar PV, Estrada-Franco JG, Navarro-Lopez R, Ferro C, Haddow AD, Weaver SC. Endemic Venezuelan equine encephalitis in the Americas: hidden under the dengue umbrella. Future Virol. 2011;6(6):721-740. doi:10.2217/FVL.11.5
[9] Blohm GM, Lednicky JA, White SK, et al. Madariaga virus: Identification of a lineage III strain in a Venezuelan child with acute undifferentiated febrile illness, in the setting of a possible equine Epizootic. Clin Infect Dis. 2018;67(4):619-621. doi:10.1093/cid/ciy224
[10] de Souza Costa MC, Siqueira Maia LM, Costa de Souza V, et al. Arbovirus investigation in patients from Mato Grosso during Zika and Chikungunya virus introdution in Brazil, 2015-2016. Acta Trop. 2019;190:395-402. doi:10.1016/j.actatropica.2018.12.019
[11] Lednicky JA, White SK, Mavian CN, et al. Emergence of Madariaga virus as a cause of acute febrile illness in children, Haiti, 2015-2016. PLoS Negl Trop Dis. 2019;13(1):e0006972. doi:10.1371/journal.pntd.0006972
[12] Aguilar PV, Robich RM, Turell MJ, et al. Endemic eastern equine encephalitis in the Amazon region of Peru. Am J Trop Med Hyg. 2007;76(2):293-298. doi:10.4269/ajtmh.2007.76.293
[13] Carrera JP, Cucunubá ZM, Neira K, et al. Endemic and epidemic human alphavirus infections in Eastern Panama; An Analysis of Population-based Cross-Sectional Surveys. bioRxiv. Published online January 10, [2020] doi:10.1101/2020.01.10.901462
[14] Powers AM. Asymptomatic or mild febrile cases of Madariaga: The base of the iceberg? Clin Infect Dis. 2018;67(4):622-623. doi:10.1093/cid/ciy225
[15] Rivera LF, Lezcano-Coba C, Galué J, et al. Clinical and epidemiological characteristics of Madariaga and Venezuelan equine encephalitis virus infections. medRxiv. Published online February 3, [2024] doi:10.1101/2024.02.02.24302220
[16] Carrera JP, Araúz D, Rojas A, et al. Real-time RT-PCR for Venezuelan equine encephalitis complex, Madariaga, and Eastern equine encephalitis viruses: application in human and mosquito public health surveillance in Panama. J Clin Microbiol. 2023;61(12):e0015223. doi:10.1128/jcm.00152-23
[17] Gil LHVG, Magalhaes T, Santos BSAS, et al. Active circulation of Madariaga virus, a member of the Eastern equine encephalitis virus complex, in northeast Brazil. Pathogens. 2021;10(8):983. doi:10.3390/pathogens10080983
[18] Hull R, Nattanmai S, Kramer LD, Bernard KA, Tavakoli NP. A duplex real-time reverse transcriptase polymerase chain reaction assay for the detection of St. Louis encephalitis and eastern equine encephalitis viruses. Diagn Microbiol Infect Dis. 2008 ;62(3):272-279. doi:10.1016/j.diagmicrobio.2008.07.004
[19] Lambert AJ, Martin DA, Lanciotti RS. Detection of North American eastern and western equine encephalitis viruses by nucleic acid amplification assays. J Clin Microbiol. 2003;41(1):379-385. doi :10.1128/JCM.41.1.379-385.2003
[20] Kang X, Li Y, Liu H, et al. A duplex real-time reverse transcriptase polymerase chain reaction assay for detecting western equine and eastern equine encephalitis viruses. Virol J. 2010;7(1):284. doi:10.1186/1743-422X-7-284
[21] Zink SD, Jones SA, Maffei JG, Kramer LD. Quadraplex qRT-PCR assay for the simultaneous detection of Eastern equine encephalitis virus and West Nile virus. Diagn Microbiol Infect Dis. 2013;77(2):129-132. doi:10.1016/j.diagmicrobio.2013.06.019
[22] Soria ME, Cortón M, Martínez-González B, et al. High SARS-CoV-2 viral load is associated with a worse clinical outcome of COVID-19 disease. Access Microbiol. 2021;3(9):000259. doi:10.1099/acmi.0.000259
[23] Tan L, Kang X, Ji X, et al. Validation of predictors of disease severity and outcomes in COVID-19 patients: A descriptive and retrospective study. Med (N Y). 2020;1(1):128-138.e3. doi:10.1016/j.medj.2020.05.002
[24] Granados A, Peco A, McGeer A, Gubbay JB. Influenza and rhinovirus viral load and disease severity in upper respiratory tract infections. J Clin Virol. 2017;86:14-19. doi:10.1016/j.jcv.2016.11.008
[25] Sterling TR, Vlahov D, Astemborski J, Hoover DR, Margolick JB, Quinn TC. Initial plasma HIV-1 RNA levels and progression to AIDS in women and men. N Engl J Med. 2001;344(10):720-725. doi:10.1056/NEJM200103083441003
[26] Hyde JL, Chen R, Trobaugh DW, et al. The 5’ and 3' ends of alphavirus RNAs--Non-coding is not non-functional. Virus Res. 2015;206:99-107. doi:10.1016/j.virusres.2015.01.016
[27] Sánchez-Seco MP, Rosario D, Quiroz E, Guzmán G, Tenorio A. A generic nested-RT-PCR followed by sequencing for detection and identification of members of the alphavirus genus. J Virol Methods. 2001;95(1-2):153-161. doi:10.1016/s0166-0934(01)00306-8
enviar
suscripperonal
Información
Idioma





